INTRODUCTION
복막투석 복막염은 복막투석을 받는 환자의 주요 부작용으로, 복막투석의 장기적인 성공을 위해 신속한 진단과 적절한 치료가 매우 중요하다[1]. 미생물 검사실에서는 병인균의 검출율을 높이기 위하여 통상적인 복막투석액 배양법에 원심분리, 백혈구 용해 및 자동화된 혈액배양 시스템 등을 적용하고 있지만, 복막투석액 내에 존재하는 세균의 양이 매우 적고, 배양이 되지 않거나 까다로운 균, 항균제 치료 후 등의 경우에는 종종 통상적인 배양법으로는 검출이 어렵다.
최근에는 세균 검출용 16S rRNA와 진균 검출용 internal transcribed spacer (ITS)을 표적으로 PCR과 염기서열분석을 적용하는 방법이 소개되었지만, 배양의 민감도(82.2%)에 비해 PCR의 민감도(88.9%)는 큰 차이가 없는 것으로 보고되었다[2]. 16S rRNA와 ITS 염기서열분석은 통상적인 방법으로 동정이 어려운 세균을 동정하는데 도움이 되지만, 증폭 염기서열의 위치, 크기, 균종간 유전자 일치도가 높은 균종에서의 변별력 한계 등의 제한점이 있다[3].
차세대 유전자 분석(next-generation sequencing, NGS)의 등장은 임상미생물 검사 영역에서도 많은 변화를 가지고 왔다[4]. 원내 외에서 집단 발생하는 감염의 병인균 typing 및 항균제내성이나 병독성에 관련된 유전자 검출에 NGS를 이용할 수 있고, 배양이 까다로운 세균도 검출이 가능하다[5]. 지금까지 해외 연구에서 요, 혈액, 및 정형외과 검체를 이용한 보고들이 있지만[6-8], 복막투석액에 적용한 연구는 아직까지 없다. 본 연구에서는 복막투석으로 발생한 복막염 진단에 NGS을 적용하여, 이의 진단적 유용성을 평가하여, 투석 환자의 진료에 도움이 되고자 한다.
MATERIALS AND METHODS
2018년 4월 한달 간 복막투석 복막염이 의심되어 복막투석액 배양을 의뢰한 총 21명을 대상으로 하여, 통상적인 배양법과 16S rRNA 유전자를 표적으로 하는 NGS 결과를 비교하였다.
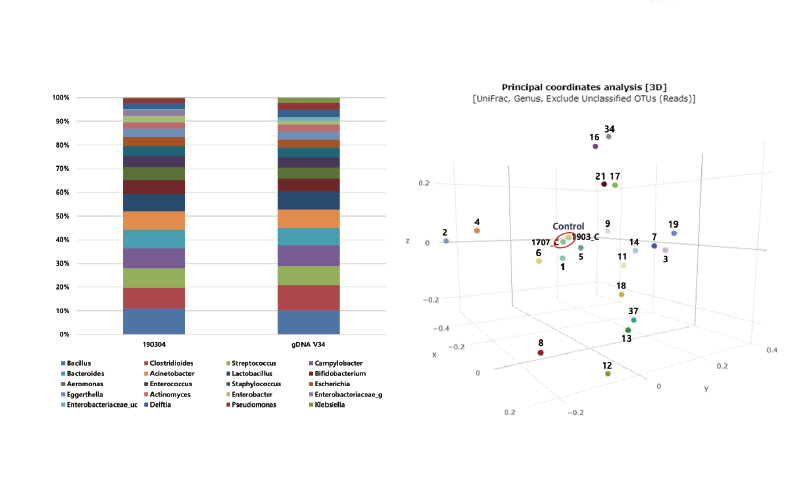
복막투석액 50 mL를 원심분리하여 얻은 침사를 초콜렛과 혈액한천배지에 획선하여 37℃, 5% CO2 배양기에서 48시간 배양하였고, 나머지 침사는 thioglycollate에 접종하여 1주일까지 배양하였다. 균 증식이 관찰되는 경우 순 배양 집락을 이용하여 MALDI Biotyper Systems (Bruker Daltonik, Bremen, Germany)으로 동정하였다. NGS 분석은 -70°C에 보관하였던 복막투석액 침사를 이용하였다. Fast DNA Spin Kit for Soil (MP Biomedicals™, Fountain Parkway, CA, USA)를 이용하여 DNA를 추출 후, 제조사의 지침에 따라 MiSeq (Illumina, San Diego, CA, USA)를 이용하여 16S rRNA 유전자의 V3, V4를 증폭하였다. 자세한 프로토콜은 를 참고하였다. Metagenomic 분석은 EzBioCloud 16s-based MTP (ChunLab, Inc, Seoul, Korea)를 이용하여 속 (genus) 수준으로 수행하였다. NGS를 이용한 균종 확인은 속 (genus) 수준으로 하였다. 정도관리를 위하여 매 run마다 음성과 양성 control을 포함하였고, 모든 검체 data와 매 run 마다 시행한 gDNA Mock V34 (1707_C)과 MiSeq QC (1903_C) 양성 control의 data를 함께 principal coordinate analysis로 분석하여 run간의 변이(variation)가 허용 범위 내에 드는지 확인하였다(Fig. 1). 복막투석 복막염의 발생과 16S rRNA 표적 NGS시 microbiome의 다양성과 연관이 있는지 알아보기 위하여 α-diversity을 분석하였다. 전자의무기록을 검토하여 대상환자의 진성 복막감염 유무, 복막염 최종 치료 항균제, 치료 10일째 회복(clear up), 복막투석 카테터 제거 및 치료 4주 이내 같은 균주에 의한 재발(relapse) 등의 임상적인 특징을 파악하였다.
RESULTS
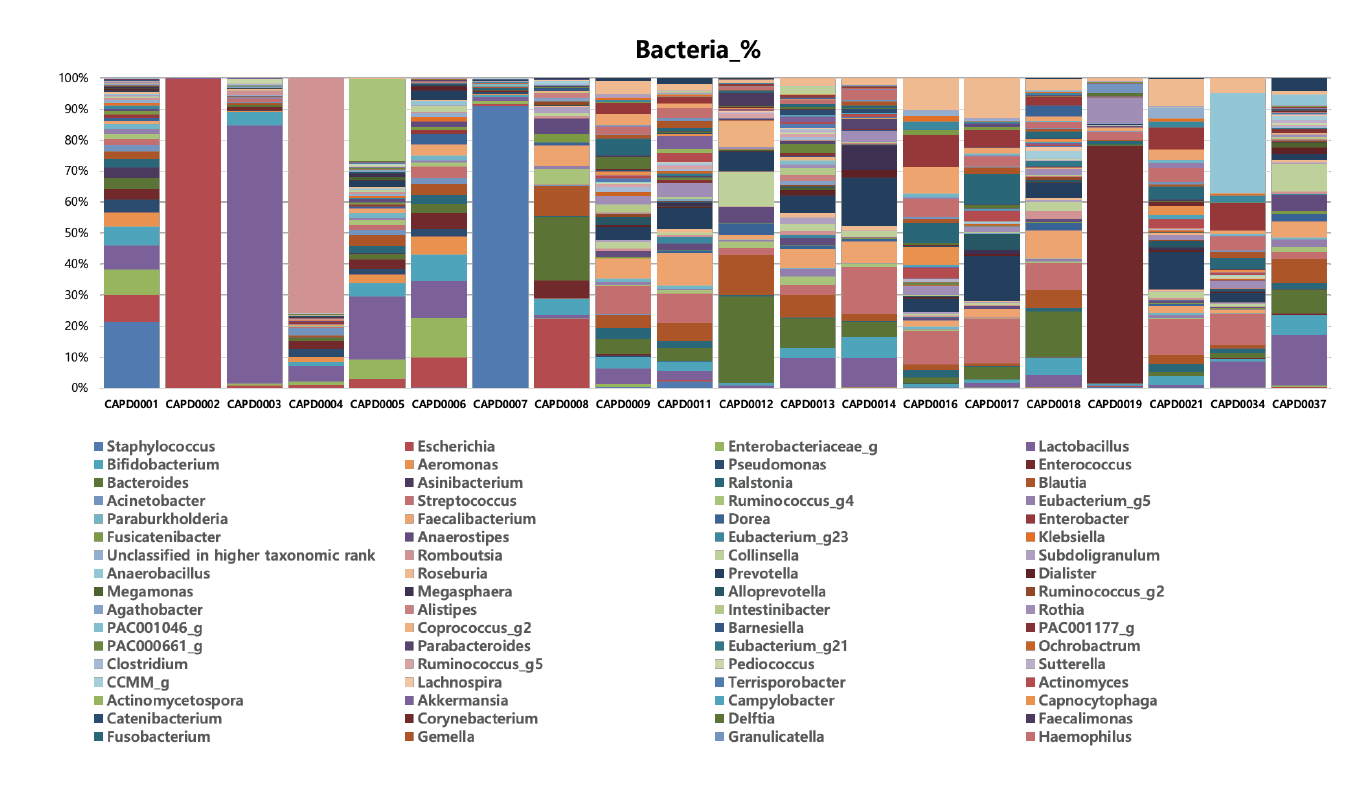
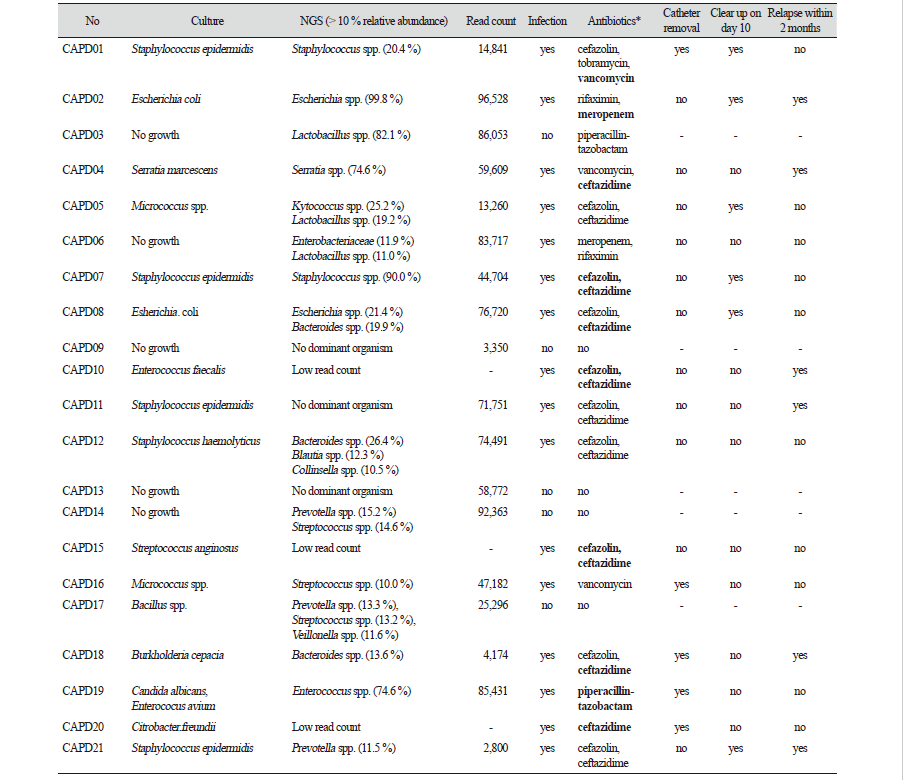
총 15개 복막투석액 검체에서 다양한 세균이 16Sr RNA 표적 NGS로 검출되었으며, relative abundance cut off 1%로 분석하였을 때 복막투석액 내 microbiome은 1-29 종류의 속(genus)이 검출되었다(Fig. 2). 3개의 검체(CAPD10, CAPD15, CAPD20)는 low read count로 분석할 수 없었고, 3개의 검체(CAPD09, CAPD11, CAPD13)는 relative abundance 10%를 초과하는 우세균이 없었다(Table 1).
총 16개의 배양 양성 검체 중 6개(CAPD01, CAPD02, CAPD04, CAPD07, CAPD08, CAPD19)의 검체에서 NGS로 통상적인 CAPD 배양에서 분리된 균주와 동일한 속(genus) 의 균주를 검출하였다(Table 1). 총 5개의 배양 음성 검체 중 NGS로 3개 검체에서 균을 검출 할수 있었는데, Lactobacillus spp. (CAPD03, CAPD06), Enterobacteriaceae (CAPD06), Streptococcus spp. (CAPD14) 및 Prevotella spp. (CAPD14)를 검출하였다. 배양 양성 검체 중 5개 검체에서 NGS로 혐기성 세균을 추가로 검출하였는데, Bacteroides spp. (CAPD08, CAPD12, CAPD18), Blautia spp. (CAPD12), Prevotella spp. (CAPD17, CAPD21) 및 Veillonella spp. (CAPD17)이었다(Table 1).
연구 대상 환자 21명 중 16명을 임상의가 진성 복막감염(true peritonitis)으로 판단하였는데, 이 중 한 예(CAPD06)가 배양 음성 복막염이었고, NGS로 Lactobacillus spp. 와 Enterobacteriaceae 를 검출하였다. 진성 복막염 환자들은 적절한 항균제 치료를 받았고, 이 중 5명은 catheter를 제거하였다. 2개월 이내 재발한 6예(CAPD02, CAPD04, CAPD10, CAPD11, CAPD18, CAPD21)는 재발 시점에서 시행한 복막투석액 배양에서 최초 배양에서 분리된 균종과 동일한 균주를 재검출하였다(Table 1). 임상의가 복막감염이 아닌 것으로 판단하였으나, 항생제를 투약한 한 환자(CAPD03)는 검체 채취일부터 piperacillin-tazobactam을 투여하였으나, 배양결과가 음성임을 확인 후 투약을 중단하였다.
복막투석 복막염의 발생과 16 Sr RNA 표적 NGS시 microbiome의 다양성과 연관이 있는지 알아보기 위하여 분석이 가능했던 19 검체의 α-diversity와의 연관성을 조사하였다. Shannon (P = 0.7668), Simpson (P = 0.3988), OTUs (P = 0.9632), ACE (P = 0.6131), Chao1(P = 0.8899) 같은 다양한 α-diversity지표들을 진성 복막염과 임상의의 판단으로 감염이 의심되지 않는 경우 간에 비교한 결과, Wilcoxon rank sum test 수행한 P 값은 모두 > 0.05 이상으로 두 군 간에 차이가 없었다.
DISCUSSION
복막염의 진단 기준으로 적합한 임상양상(복통), 복막투석액 내 50% 이상의 다형핵구와 100 μ/L을 초과하는 백혈구 관찰 및 미생물 배양 양성의 3개의 조건 중 2가지 이상을 흔히 사용한다[9]. 복막염의 병인균 검출을 위해 배양을 실시하면 약 13%은 배양 음성이고, 11%은 두 종 이상의 균이 원인이며(polymicrobial), 단일 세균이 분리된 경우 그람 양성균이 대부분(53.4%)을 차지한다는 보고가 있다[10]. 임상적으로 복막염이 의심되는데 배양이 음성인 경우 적절한 항생제를 결정하기 어렵기 때문에 문제가 되며, mycobacteria, 진균 등의 비정형 세균들에 의한 경우가 많다[11].
미생물 배양은 적절한 항균제 선택을 위하여 반드시 필요한 자료를 제공하지만, 배양에 소요되는 시간이 길고, 최근 항균제를 투여받았거나, 세균 수가 매우 적은 경우 및 잘 자라지 않는 균에서는 만족할 만한 자료를 얻기가 어렵다. 지금까지 NGS는 비용과 검사의 난이도가 높아 통상적인 미생물 검사에 사용하는데 적합하지 않았지만, 사용자 편의성이 높아진 장비들의 개발, 저렴해진 검사 단가, 사용이 편리한 상품화된 생물정보학(bioinformatics) 도구의 보급 등의 덕분으로 앞으로는 통상적인 미생물 검사업무에서 감염질환의 병인균을 규명하는 등의 목적으로 NGS를 사용하는 것이 더 이상 먼 미래의 일이 아니다 [12].
지금까지 임상검체에서 NGS를 적용한 예를 살펴보면, 요로감염이 의심되는 환자에서 얻은 60개의 요 검체, 23개의 혈액배양 양성병 및 21개의 조직, 체액, 관절관액 등의 정형외과 검체에서 16S-23S rRNA NGS를 적용하였다[6]. 이 보고에 의하면 NGS법이 16S rRNA 유전자의 PCR-sequencing 보다 균종의 변별력이 더 좋았고, 소변배양과 혈액배양에서 통상적으로 검출되는 병인균을 신속하고 정확하게 동정하였고, 정형외과 검체에서 병인균을 좀 더 민감하게 검출할 수 있었다[6]. 특히 패혈증 환자에서 병인균의 신속한 검출은 매우 중요한데, NGS를 이용하여 검체 준비부터 보고까지 30시간 이내로 가능하였다[7]. 소아 면역저하환자의 혈액에서 NGS를 시행하여 병인균을 검출한 보고에서는 12명의 혈류감염 환자의 혈액에서 NGS로 검출한 우세균(dominant bacteria)이 혈액배양 결과와 일치한 경우가 8명이었으며, 도관관련 혈류감염(catheter-related bloodstream infection) 환자 2명에서는 패혈증의 병인균을 발병 7일 이전에 미리 검출할 수 있었다[8].
최근에는 16Sr RNA 유전자만 대상으로 NGS를 실시하는 것이 아니고 검체 내 존재하는 숙주(host)를 포함 모든 DNA에 대해 NGS를 실시하여 분석하는 metagenomic analysis도 소개되고 있다[13]. Metagenomic analysis를 적용하려면 background DNA를 제거하는 적절한 알고리즘의 적용이 필요하고, 관절강액같은 무균 검체에 주로 적용이 가능하다는 제한점이 있지만, 균종 정보 외에도 병독성 인자나 항균제 내성균 같은 정보도 추가로 얻을 수 있는 장점이 있다.
본 연구에 NGS로 복막투석액에서 다양한 세균을 검출할 수 있었는데, 일부 균주들은 그 의미를 해석하기 어려웠다. Lactobacillus가 검출된 경우 오염균일 가능성이 높지만, L. casei, L. paracasei, L. acidophilus, 및 L. rhamnosus에 의한 복막 투석 복막염이 드물게 보고된 적이 있어[14-17], Lactobacillus spp.를 병인균에서 완전히 배제하기는 어렵다. Kytococcus, Blautia spp. 및 Collinsella spp. 모두 복막투석 복막염의 병인균이라는 보고는 아직 없다. Kytococcus spp.는 methicillin에 내성을 보이는 그람 양성 구균으로 Genus Micrococcus에서 분화된 균종이며[18], 그 역할은 잘 알려져 있지 않지만 Blautia spp.는 정상인의 장내 metagenome 분석에서 흔히 분리되는 것으로 보고되어 있다[19]. Collinsella spp.는 장내 microbiota로 알려져 있다[20].
대부분의 검사실에서 통상적인 배양에서 혐기성 세균의 검출에 적합한 배지와 조건을 적용하고 있지 않은데, 그 이유로 복막투석액은 산소포화도가 높아 혐기성 세균은 잘 자라지 않을 것으로 생각하기 때문이다. 하지만 NGS로는 많은 검체에서 혐기성 세균을 검출할 수 있었는데, Prevotella spp.나 Veillonella spp.의 경우 임상적 의의를 판단하기 어렵지만, Bacteroides spp.경우 병독성도 높고, 이 균주에 의한 복막염에 관한보고가 있어 병인균의 가능성이 있다[21, 22]. 이 외에도 복막투석 환자에서 자동화된 혈액배양 방법을 이용하면 통상적인 배양법으로 검출되지 않았던 Bacteroides fragilis group 등의 혐기성 세균을 혐기성 혈액병에서 검출할 수 있다는 보고가 있다[23].
복막투석액 내 microbiome의 다양성의 의미에 대해 아직까지 알려진 바는 없지만, 진성 감염인 경우 병인균의 우세한 증식 및 상재균의 감소로 복막투석액 내 microbiome이 다양성이 감소할 가능성이 높다. 아쉽게도 본 연구에서 각 그룹의 α-diversity를 비교해 보았을 때, 통계적으로 유의한 차이를 얻을 수 없었는데, 분석 대상의 수가 너무 적은 것이 원인이라 생각된다.
본 연구는 복막투석 복막염 진단을 위해 NGS를 처음 적용한 것이라는 의의가 있으며, 통상적인 배양에 비해 16S rRNA 표적 NGS는 혐기성 세균과 배양 음성 검체에서 추가적인 세균 검출이 가능한 것을 확인할 수 있었다.
요약
배경: 복막염은 복막투석과 관련된 가장 흔한 합병증이며, 효과적인 치료를 위해서는 원인균의 신속 정확한 동정이 필수적이다. 차세대 염기서열분석법(NGS)을 이용하여 16Sr RNA 유전자를 표적으로 미생물을 검출할 수 있는데, 지금까지 해외 연구에서 요, 혈액 및 관절액 등 검체에서 검출이 가능하다고 알려져 있지만, 복막투석액을 이용한 보고는 아직까지 없다. 본 연구에서는 NGS를 이용하여 복막투석 복막염의 원인균 검출이 가능한지 알아보았다.
방법: 2018년 4월 한달간 복막투석 복막염이 의심되어 배양을 의뢰한 총 21명의 환자를 대상으로 통상적인 배양법과 16S rRNA 유전자 표적 차세대 염기서열분석법의 결과를 비교하였다. 임상적인 특징은 담당 주치의가 전자의무기록을 검토하여 파악하였다.
결과: 복막투석액의 microbiome은 검체마다 다양한 양상을 보였는데, 1-29 종의 genus로 구성되었다. 차세대 염기서열분석법으로 분석 가능했던 18개 검체 중 6개 검체(CAPD01, CAPD02, CAPD04, CAPD07, CAPD08, CAPD19)의 배양에서 검출된 균과 동일한 속(genus)의 균주를 확인할 수 있었다. 6개 검체에서 Bacteroides나 Prevotella같은 혐기성 균종을 추가로 검출할 수 있었고, 세균배양 음성이었던 5검체 중 3개의 검체(CAPD06, CAPD14, CAPD17)에서 병원균의 가능성이 높은 genus를 검출할 수 있었다.
결론: 본 연구는 복막투석 복막염 진단을 위해 차세대 염기서열분석법을 최초로 적용한 결과, 16S rRNA 표적 NGS는 혐기성 세균의 검출과 배양 음성 검체에서 병원균의 확인에 추가적인 도움을 줄 수 있는 것으로 생각되었다.