INTRODUCTION
Beta-lactam 항생제에 내성을 가지게 하는 광범위 베타락탐 분해효소(extended-spectrum beta-lactamase, ESBL)를 생성하는 그람음성균(Gram-negative bacilli)의 확산에 따라[1], 그람음성균 감염증의 주된 치료 항생제로 카바페넴(carbapenem)계 항생제 사용이 증가하여 카바페넴 내성 장내세균(carbapenem-resistant Enterobacteriaceae, CRE) 또한 급속도로 증가하면서 전 세계적으로 문제가 되고 있다[2,3]. 국내에서는 Escherichia coli와 Klebsiella pneumoniae에서 1% 미만의 카바페넴 내성률이 보고 되었지만[4], 최근 카바페넴 분해효소 생성 장내세균(carbapenemase-producing Enterobacteriaceae, CPE)의 분리에 대한 보고가 지속적으로 증가하고 있다[5]. CRE는 카바페넴 분해효소 비생성 장내세균(carbapenemase-nonproducing Enterobacteriaceae, CNPE)과 카바페넴 분해효소 생성 장내세균(CPE)으로 구분할 수 있다[6]. 그람음성균의 카바페넴계 항생제에 대한 내성을 획득하는 주요 기전은 카바페넴 분해효소 생성에 의한 항생제 불활성화이다. 카바페넴 분해효소를 전사하는 유전자는 플라스미드(plasmid)와 트랜스포존(transposon) 등과 같은 이동성 유전요소(mobile genetic element)에 존재하는 경우가 많아 동종간 혹은 이종간 내성의 전파가 가능하기 때문에, 실제로 의료기관이나 지역사회에서의 감염관리 및 감시의 대상이 된다[7].
카바페넴 분해효소는 Ambler의 분류법에 따라 다음과 같이 3가지로 분류된다: (i) class A; KPC (Klebsiella pneumoniae carbapenemase), GES (Guiana extended-spectrum beta-lactamase) 등, (ii) class B; NDM (New Delhi metallo-beta-lactamase), IMP (imipenemase), VIM (Verona integron-encoded metallo-β-lactamase) 등, 그리고 (iii) class D; Oxacillinase (OXA)-48-like 등[1,6]이다. Lee EJ 등의 연구결과에 따르면[8], 질병관리본부에 보고된 카바페넴 분해효소 장내세균 분리 건수는 2012년 39건이었으나, 2017년에 2,953주로 급증하였다. 카바페넴 분해효소 장내세균 중에서, KPC형이 가장 흔하였으며, 그 중에서도 KPC-2가 가장 큰 비율을 차지하였다. blaKPC-2 유전자는 2010년에 K. pneumoniae에서 처음으로 검출된 이후 가장 많이 보고 되었다[9-11]. Yoon E-J 등의 보고에서도, 국내 카바페넴 분해효소 장내세균의 분리 현황을 확인하였을 때, KPC-2 생성 K. pneumoniae가 절반 이상을 차지하였고, 두 번째는 NDM-1 생성 E. coli, 다음은 OXA-48-like 생성 E. coli가 뒤를 이었다[5]. KPC-2 생성 K. pneumoniae의 strain type (ST)을 살펴보았을 때 ST11과 ST258이 전세계적으로 가장 흔하며, 국내에서는 ST11과 ST307의 광범위한 전파가 보고된 바 있다[5,8]
본 연구에서는 2018년 4월부터 12월까지의 국내 한 종합병원에서 분리된 KPC형 카바페넴 분해효소 생성 K. pneumoniae들의 분자 역학적 연관성을 밝히고, 임상적 의의를 확인하고자 하였다.
MATERIALS AND METHODS
1. 대상 균주
2018년 국내 1개의 종합병원에서 4월부터 12월까지 임상검체에서 분리된 K. pneumoniae 균주 중 imipenem, meropenem 및 ertapenem을 포함한 카바페넴 계열 항생제에 비감수성을 보이는 균주를 수집하였다. 동일 환자에서 K. pneumoniae가 반복 분리 된 경우 최초 분리 균주를 수집하였다.
2. 균종 동정
균주의 동정은 말디토프 질량분석기(matrix-assisted laser desorption lonization-time-of flight mass spectrometer, MALDI-TOF MS) 기반의 Bruker Biotyper (Bruker, Billerica, MA, USA)를 사용하였다. 수집된 K. pneumoniae 균주를 MacConkey 배지(BANDIO, Pocheon, Korea)에 배양하여 얻은 단일균 집락을 말디토프 플레이트에 얇게 도말한 후, 매트릭스 용액을 도포하여 완전히 건조시킨 후 MALDI-TOF MS를 시행하였다. 균종 동정 점수의 기준은 2.0 이상으로 하였다.
3. 항생제 감수성 시험
카바페넴 계열 항생제 감수성 시험은 Phoenix M50 (Becton Dickinson, Franklin Lakes, NJ, USA)의 NMIC-203 패널을 이용하였다. 또한, 무작위로 선정된 대표균주를 대상으로 piperacillin, ampicillin-sulbactam, cefazolin, cefotaxime, ceftazidime, cefepime, aztreonam, cefoxitin, imipenem, meropenem, ertapenem, amikacin, gentamicin, tigecycline, colistin, ciprofloxacin, 및 trimethoprim-sulfamethoxazole에 대하여 디스크 확산법으로 항생제 감수성 시험을 시행하였다. Clinical and Laboratory Standard Institute (CLSI) 해석 기준에 따라 판독하였으며[12], 표준 균주는 E. coli ATCC 25922를 사용하였다.
4. 내성기전 분석
카바페넴 내성 K. pneumoniae 균주를 대상으로 ESBL (CTX-M-1군, CTX-M-2군, CTX-M-9군 및 CTX-M-25군), AmpC (CMY-1, CMY-2, ACC, ACT, FOX), 및 carbapenemase (KPC, IMP, VIM, NDM, GES 및 OXA-48)에 대하여 PCR 및 시퀀싱을 진행하였다. PCR 조건은 ESBL과 carbapenemase의 경우 pre-denaturation 95°C에서 5분, denaturation 94°C에서 30초, annealing 56°C에서 20초, extension 72°C에서 40초를 30사이클을 통해 증폭시킨 후 72°C에서 7분 동안 반응시켰고, AmpC는 pre-denaturation 94°C에서 5분, denaturation 94°C에서 30초, annealing 58°C에서 30초, extension 72°C에서 1분을 25사이클을 통해 증폭시킨 후 72°C에서 7분 동안 반응시켜주었다. 증폭 산물은 염기서열분석을 통해 얻어진 결과를 NCBI의 BLAST ()와 비교하여 유전형을 확정하였다. 본 연구에서 사용된 primer의 염기서열은 Table 1에 정리하였다.
Table 1
Oligonucleotide sequence of the primers used in this study
5. Pulsed field gel electrophoresis (PFGE)
K. pneumoniae의 유전체(gDNA)를 plug에 고정시켜서, XbaI (TaKaRa biotechnology, shiga, Japan) 제한효소를 처리하고, GenePath System (Bio-rad Laboratories, Hercules, CA, USA)에 6 V/cm, switch time 2.1초 11°C 조건으로 전기영동하였다. 동일 pulsotype의 기준은 다음과 같이 정하였다: (1) PFGE banding pattern 상 3개의 band 미만의 차이를 보이는 경우, 그리고 (2) 1.0% Dice 계수(coefficient) 산술 평균(arithmetic averages)을 이용하여 unweighted pair group에 기초로 생성된 계통수의 band 유사성을 InforQuestFP Version 4.5 (Bio-Rad, Hercules, CA, USA)로 분석하여 80% 이상인 경우[19].
6. Multilocus sequence typing (MLST)
PFGE 밴드유형의 유사성을 기준으로 대표적인 pulsotype 을 확인하고, 각각의 pulsotype에 대해 대표적인 1균주를 무작위 선별하여 7개의 housekeeping 유전자인 rpoB, gapA, mdh, pgi, phoE, infB, tonB을 대상으로 PCR 및 염기서열분석을 진행하였으며, 분석된 염기서열을 K. pneumoniae MLST database (http://bigsdb.pasteur.fr/klebsiella/klebsiella.html)와 비교하여 allelic type과 sequence type을 확인하였다.
7. 임상정보 조사
KPC 생성 K. pneumoniae가 분리된 환자들의 연령, 성별, 분리된 검체의 종류, 입원 경로, 입원 병동, 예후 등에 대하여 조사하였다. 전원된 환자의 경우 전원된 병원의 종류(1차 의료기관, 요양병원, 종합병원)를 확인하였다. 또한, 입원환자의 경우 입원 병실의 종류(일반병실 또는 중환자실)을 확인하였다.
RESULTS
1. KPC-2 생성
총 100주의 KPC-2 생성 K. pneumoniae가 분리되었다. KPC-2 생성 K. pneumoniae는 2018년 4월에 1주 분리되었으나, 5월에는 6주, 6월, 7월에는 각각 13주가 분리되었으며, 8월에 25주로 가장 많이 분리되었다. 이후 9월에는 17주로 감소하였으며, 10월에 8주, 11월에 11주, 그리고 12월에 5주로 서서히 감소하였다(Fig. 1).
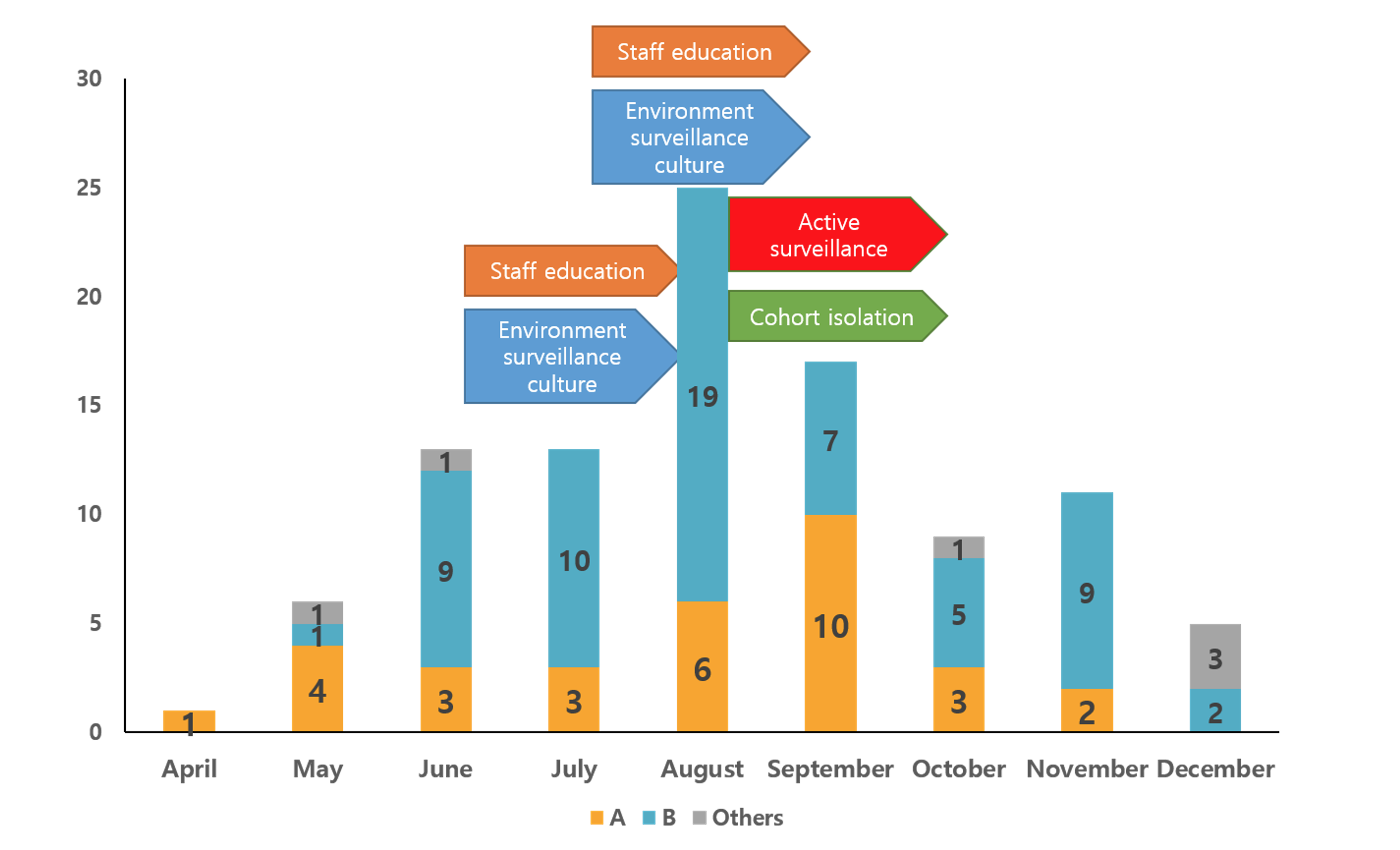
Figure 1. Monthly isolation of KPC-2 producing K. pneumoniae isolates stratified by PFGE banding patterns. Bar graphs indicate the numbers of KPC-2 producing K. pneumoniae isolates collected in each nine month in 2018 and arrows indicate the activity performed for eradicating a KPC-type carbapenemase producing K. pneumoniae isolates outbreak. Yellow bar, pulsotype A; Blue bar, pulsotype B; Gray bar, other pulsotypes.
KPC-2 생성 K. pneumoniae가 분리된 환자 중 남성은 56명(56.0%), 여성은 44명(44.0%)으로 남성환자가 여성환자보다 많았다. 환자의 연령은 0세부터 96세까지였으며 평균나이는 67세였고, 0세, 9세 환자 각각 1명을 제외한 모든 환자는 20대 이상의 성인이었다. 특히, 78명의 환자가 60세 이상으로 노인 환자의 비율이 높았다. 전체 환자 중 21.0% (n = 21)가 외부 병원에서 전원된 환자이며, 그 중 종합병원에서 전원된 환자가 42.9% (9/21), 요양병원에서 전원된 환자가 28.6% (6/21), 의원에서 전원된 환자가 28.6% (6/21)였다. 검체의 유형은 호흡기 검체 분리 균주가 37.0% (n = 37)로 가장 많았으며, 요 분리주 22.0% (n = 22), 농양 분리 주 15.0% (n = 15) 순으로 많았다(Table 2).
Table 2
Patient characteristics
병동 별로는 KPC-2 생성 K. pneumoniae는 일반병동에서 77.0% (n = 77)가 분리되어 가장 분리 빈도가 높았으며, 중환자실에서 21.0% (n = 21), 외래에서 2.0% (n = 2)가 분리되었다. 단일 병동은 중앙 중환자실에서 17.0% (n = 17)로 가장 많이 분리되었으며, 64병동(15.0%, n = 15), 63병동(13.0%, n = 13)이 뒤를 이었다. KPC-2 생성 K. pneumoniae 분리 환자의 입원기간 내 사망률은 37.0% (n = 37)였으며, 30일내 사망률은 25.0% (n = 25)였다.
2. KPC-2 생성
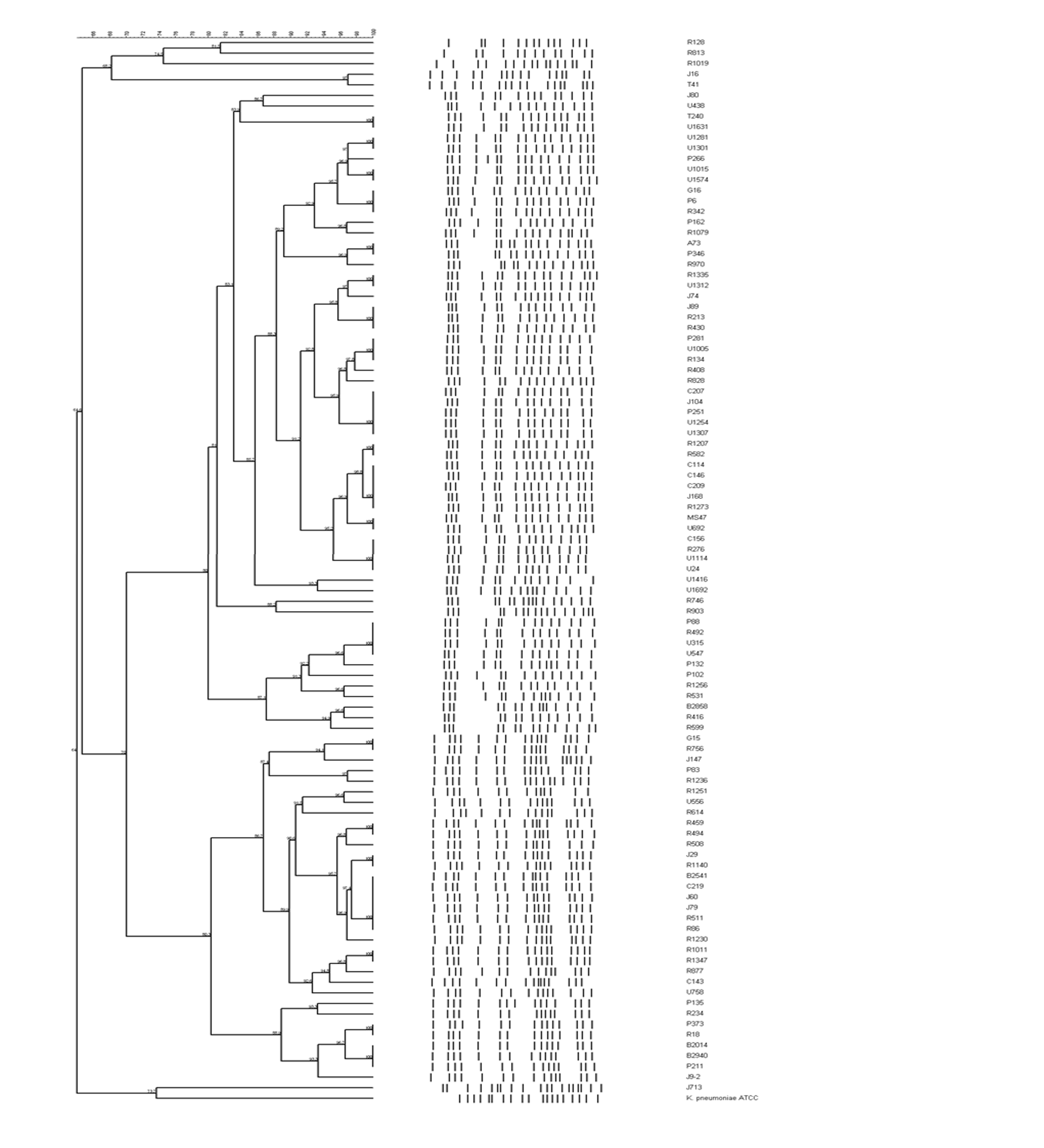
KPC-2 생성 K. pneumoniae 분리 균주들을 80% 이상의 PFGE 클론 유사성을 기준으로 분석하였을 때, 2가지 pulsotype이 94.0% (pulsotype A, 31%, n = 31; pulsotype B, 63.0%, n = 63)를 차지하였고, 그 외 6 개의 다른 pulsotype이 확인되었다. 두 가지 주요한 pulsotype A, B 사이의 클론 유사성은 약 70% 였다(Fig. 2).
Pulsotype A에 속하는 K. pneumoniae 균주들은 Phoenix system (Becton Dickinson)으로 항생제 감수성 시험을 시행하였을 때, 모두 ampicillin, piperacillin-tazobactam, cefazolin, ceftazidime, cefotaxime, aztreonam, ertapenem, meropenem, 및 levofloxacin에 내성 표현형을 보였으며, 1주를 제외한 모든 균주가 gentamicin과 amikacin에 감수성 표현형을 보였다(Table 3). 디스크 확산법에 의한 감수성 확인 시험에서 Phoenix system (Becton Dickinson)의 결과와 유사한 결과를 보여 주었으며, MLST 분석법에 따라 ST395 (rpoB-gapA-mdh-pgi-phoE-infB-tonB, 3-1-2-4-1-1-4)에 해당하였다(Fig. 3). 또한, blaKPC-2 이외에 blaCTX-M-15 유전자가 확인되었으며, 그 이외의 다른 ESBL을 전사하는 유전자 또는 AmpC β-lactamase는 확인되지 않았다. Pulsotype B에 속하는 K. pneumoniae 균주들은 모든 beta-lactam 항균제에 대하여 내성 표현형을 보였으며, 2주를 제외한 모든 균주에서 gentamicin 동반내성을 보여주었다. 디스크 확산법에 의한 감수성 확인 시험에서 Phoenix system (Becton Dickinson)의 결과와 유사한 결과를 보여 주었으며, ST307 (4-1-2-52-1-1-7)에 해당하였다(Fig. 4). Pulsotype B를 보이는 K. pneumoniae 균주들 역시 blaCTX-M-15을 동시에 가지고 있었다.
Table 3
Antimicrobial susceptibility results of KPC-producing K. pneumoniae isolates obtained by Phoenix system

Figure 3. Representative results of disk diffusion method for antimicrobial susceptibility testing in KPC-2 producing K. pneumoniae isolates exhibiting pulsotype A (ST395). (A) shows results of zone diameter for 12 antibiotics on MH 150 Ø agar, (B) shows results of zone diameter for remaining 6 antibiotics on MH 90 Ø agar, and (C) is results of antimicrobial susceptibility. R, resistant; I, intermediate; S, susceptible.

Figure 4. Representative results of disk diffusion method for antimicrobial susceptibility testing in KPC-2 producing K. pneumoniae isolates exhibiting pulsotype B (ST307). (A) shows results of zone diameter for 12 antibiotics on MH 150 Ø agar, (B) shows results of zone diameter for remaining 6 antibiotics on MH 90 Ø agar, and (C) is results of antimicrobial susceptibility. R, resistant; I, intermediate; S, susceptible.
DISCUSSION
CPE는 원내 감염 및 지역 사회의 항생제 내성 전파에 가장 중요한 다제 내성균 중 하나로서, 2017년 질병관리본부의 통계에 따르면 국내에서 2014년 175건, 2015년 565건, 216년 1,453건, 2017년 2,657건으로 점차 증가되고 있는 것으로 보고되었고[8], CPE 중에서도 KPC 생성 K. pneumoniae가 가장 많았다. 실제로, 본 연구의 균주 수집 기간 동안에 KPC형 이외의 다른 유형의 카바페넴 분해효소를 생성하는 그람음성균은 분리되지 않았다.
본 연구에서, 2가지의 pulsotype이 94% (pulsotype A, 31%, n = 31; pulsotype B, 63.0%, n = 63)를 차지하였고 PFGE 패턴 80%이상 일치하였다. 두 가지 주요 pulsotype의 균주들은 amikacin 또는 gentamicin을 제외한 대부분의 베타락탐계 항생제에 내성을 보이는 다제내성 표현형을 보였다. 따라서, 두 종류의 다제 내성(multidrug-resistant) K. pneumoniae 클론(ST395, ST307)에 의한 병원 내 집단발생이 일어났음을 나타낸다. ST307은 2014년 국내에서 처음 발생한 후, 2014년 14건에서 2015년에는 82건으로 국내 보고가 있었던 클론이었으며[5], ST395는 국내에서의 아직 보고가 없었지만 이탈리아에서 보고된 적이 있다[13]. 하지만, 본 연구에서는 pulsotype 별로 1주만을 임의 선정하여 MLST를 시행하였기 때문에 나머지 각 pulsotpye 내 균주들이 동일한 ST에 포함되지 않을 가능성이 있지만, PFGE 밴드 유사성이 80% 이상이므로 같은 clonal complex에 포함될 것으로 판단된다.
100명의 KPC-2 생성 K. pneumoniae 분리 환자 중 78명의 환자가 60세 이상의 노인 환자였고, 많은 문헌에서 보고된 것처럼 노인 환자가 감염증에 취약한 것으로 여겨진다. 남성환자가 여성환자보다 많았으나, pulsotype 별 성별의 차이는 통계적으로 유의미하지 않았다(P = 0.371). 객담 검체가 37%로 가장 많이 분리되었는데, 이는 다제내성균의 보균 또는 군집(colonization)이 폐렴 등의 호흡기 관련 감염의 원인이 되었다는 것을 보여주었다. KPC-2 생성 K. pneumoniae가 분리된 환자들의 병동을 살펴봤을 때 단일병동 중에서도 중앙 중환자실에서 17%로 가장 많이 분리되었다. 중환자실 입원환자는 KPC-2 생성 K. pneumoniae에 감염의 위험인자로도 작용한다는 많은 보고가 있다[14-17]. 중환자실 입원환자의 경우 면역력이 저하된 환자의 비율이 높고 질병의 중증도가 높기 때문에 다제내성균 감염의 위험이 크며, 따라서, 감염관리 활동의 주요 대상이 되어야 한다.
KPC-2 생성 K. pneumoniae 분리 환자의 입원 내 사망률은 37%였고, 30일 이내 사망률은 25%로 매우 높았다. 이탈리아에서 2010년부터 2013까지 5개의 종합병원에서의 KPC 생성 K. pneumoniae 의 감염 환자의 예후를 살펴보았을 때 총 661명의 환자 중 225명(34.1%)의 환자가 14일 이내 사망하였고 그 중 217명의 환자가 3가지 이상의 항생제를 동시 투약을 한 것으로 보고되었다. 이러한 결과로 볼 때, 다른 균과 달리 CPE 감염 시 다제내성 표현형으로 인해 적절한 항생제 사용이 늦어지고 이는 높은 사망률과 이어지는 것으로 볼 수 있다[18].
본 연구의 KPC-2 생성 K. pneumoniae 분리에서, 4월 첫 발생 후 점점 증가하여 8월에는 월간 분리건수가 25건으로 가장 많았으며, 이 후 점차 감소하여 12월에는 5건으로 줄었다. CPE 분리 건수 증가로 병원에서는 직원 대상 감염관리 교육 및 환경배양을 7월, 8월에 시행하였으며, 9월에는 다제내성균 보균 위험성이 큰 환자군을 대상으로 능동 감시 및 보균 환자를 대상으로 코호트 격리를 시작하였으며, 이 후 분리 건수가 현저히 감소되었다. 다제 내성균의 원내 전파 예방을 위한 감염관리 활동이 중요함을 나타낸다(Fig. 1).
다제내성균에 대한 감시 및 전파예방을 위한 감염관리 활동은 병원 입원환자의 사망률을 줄이는데 필수적인 요소이다. 본 연구에서 KPC-2 생성 K. pneumoniae 집단발생(outbreak)의 임상정보 및 감염 요인을 조사하였으며, 두 가지 클론성의 KPC-2 생성 K. pneumoniae를 확인하였고, 이들 균주는 다른 계열의 항생제에도 동시내성을 보였다. 또한 감염환자의 사망률도 매우 높아, 다제내성균의 전파 예방을 위한 감염관리 활동의 중요성을 확인하였다. 따라서, 다제내성균의 병원 내 집단발생을 예방하기 위하여, 병원내 전파 클론의 빠른 확인 및 적절한 감염관리 활동이 필요하다.
요약
배경: Carbapenemase-producing Enterobacteriaceae (CPE)는 전세계적으로 빠르게 증가하고 있고, 특히 KPC-2 생성 Klebsiella pneumoniae는 높은 비율을 차지하고 있어 공중보건의 큰 위협으로 자리 잡고 있다. 본 연구는 국내 한 종합병원에서 발생한 KPC-2 생성 K. pneumoniae 균주 간의 분자 역학적 연관성을 확인하여 임상적 의의를 확인하고자 한다.
방법: 2018년 4월부터 12월까지 임상에서 분리된 carbapenem계 항생제에 비감수성인 K. pneumoniae를 수집하여 본 연구에 사용하였다. 수집된 균주 중 pulsotype별 대표 균주를 무작위로 선정하였으며, 선정된 균주를 대상으로 디스크 확산법을 통해 항생제 감수성 여부를 확인하였으며, 중합효소연쇄반응 및 시퀀싱으로 KPC형 생성 여부를 확인하였다. 또한, pulsed-field gel electrophoresis (PFGE)와 multilocus sequencing typing를 통해 분자역학적 연관성을 확인하였다.
결과: 총 100주의 KPC-2 생성 K. pneumoniae가 분리되었고, PFGE 결과 대표적으로 2가지의 pulsotype으로 구분되었다(pulsotype A, 31.0%, n = 31; pulsotype B, 63.0%, n = 63). Pulsotype A는 ST395에 해당하였고 pulsotype B는 ST307에 해당하였다.
결론: KPC-2 생성 K. pneumonia 집단발생 예방을 위해 임상정보 및 세균학적 요인을 조사하였으며 감염 시 높은 사망률을 갖고 있는 다제내성균 전파의 예방을 위한 적절한 감염관리 활동과 구축이 필요함을 알 수 있었다.